
シングルセルシークエンシングライブラリ作製キットSingleron

概要
穏やかなシングルセル分割用に設計されたSCOPE-chip ®マイクロウェルシステムは重力を利用して細胞を個々のマイクロウェルに誘導し、敏感な細胞や壊れやすい細胞でも安定したパフォーマンスを発揮します。
テクノロジーのハイライト
SCOPE-chipテクノロジーを用いたキットは、シングルセルの分割、細胞のバーコーディングから核酸の増幅およびライブラリの構築まで、シングルセルサンプルの処理ワークフロー全体をカバーします。
敏感な細胞タイプに偏りのない細胞処理
世界中の2900以上のラボで信頼されているパフォーマンス
チップあたり3,000~30,000個の細胞または核を捕捉
低いダブレット率
機器不要のワークフローまたは完全自動化ソリューションでワークフローを合理化
- 1. シングルセル分割
- 細胞はポアソン分布に従ってチップ上の数十万個のマイクロウェルに分割されます。
- 2. バーコーディング
- チップのマイクロウェルにバーコードビーズを追加します。
- 3. 細胞溶解とmRNA捕捉
- 細胞溶解後、固有の細胞バーコードを持つバーコードビーズがmRNAのPoly(A)テールに結合してmRNAを捕捉します。
- 4. ビーズ回収
- ピペットを用いてバーコードビーズを回収(手動システム)もしくは、は自動的に回収(自動システム)。
- 5. ライブラリ生成
- 逆転写(RT)、増幅、ライブラリ構築を含む残りのライブラリ準備手順は、標準的なエッペンドルフチューブで行われます。
- 6. シークエンシング
- シークエンシングライブラリはイルミナ社シークエンサーでシークエンシング可能です。

SCOPE-chip
Singleronのマニュアル操作用シングルセルRNAライブラリ調製キットで用いられているチップです。

Matrix NEO™
シングルセル処理の自動化
この装置は、チッププライミング、細胞分割、溶解、mRNAキャプチャをワンクリックで自動実行可能です。

システムのハイライト
最大4つのSCOPE-chip®を自動処理
簡単なワークフローと直感的な操作
信頼性が高く一貫した結果
パフォーマンスデータ
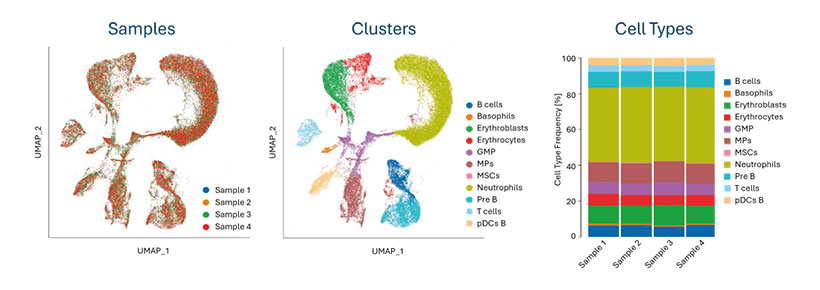
マウス骨髄の4リプリケートを、4個のSCOPE-chip ®を用いMatrix NEO™上で同時処理しました。Matrix NEO™は、シングルセルワークフローの最も時間のかかるステップを自動化し、再現性のある結果を実現します。
技術仕様
| 寸法 | 360mm×260mm×230mm |
|---|---|
| 重さ | 10kg |
| 容量 | 1~4サンプル/ラン |
| 時間 | 38分/ランニング |
GEXSCOPE® Single Cell RNA-seqライブラリ調製用キット
GEXSCOPE® Single Cell RNAライブラリ調製用キットにより、マイクロウェルベースのSCOPE®テクノロジーを使用して、生物学的サンプルの包括的なシングルセルシークエンスのためのライブラリ調製が可能となります。
特長
マイクロウェルを用いることで、希少な細胞でも高スループット処理を実現します。
機器不要なキットと専用機械用キットの2種類をラインナップ
サンプルからライブラリまで1日以内
サンプルあたり最大30,000個の細胞を捕捉
穏やかで頑健なマイクロウェルベースのシステム
GEXSCOPEシングルセルRNAライブラリキット(HD)で処理されたマウス胚のハイスループットシングルセルシークエンス。42,641個のマウス胚由来細胞のトランスクリプトーム解析により、全細胞のわずか0.15%を占める希少な細胞集団が特定されました。
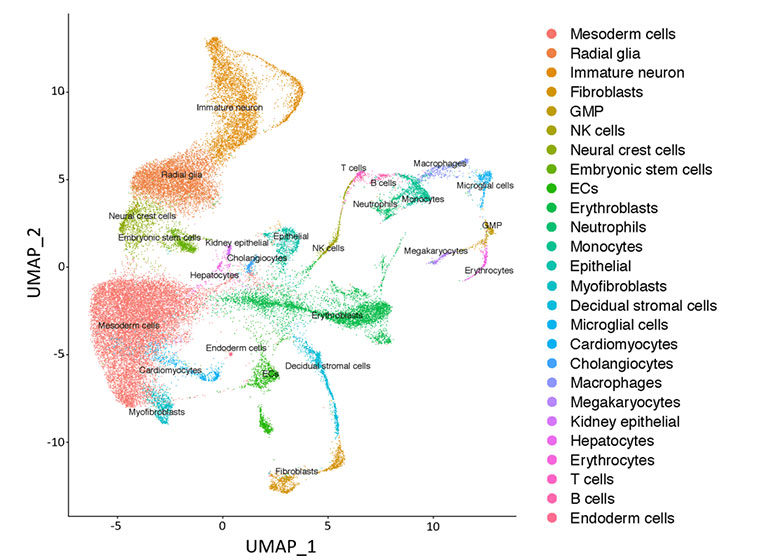
マウス胚組織から得られた42,641個のシングルセルのUMAPプロット
仕様
| 対応細胞数 | スタンダードチップ3,000~10,000 HDチップ9,000~30,000 |
|---|---|
| 生成ライブラリ | イルミナ用ライブラリ |
| ワークフロー | 6~8時間 |
GEXSCOPE®シングル核RNA-seqライブラリ調製用キット
GEXSCOPE®シングル核RNA-seqライブラリ調製用キットは凍結細胞や組織分散が困難なサンプルに対して有効なキットです。
特長
凍結細胞、大きな細胞、複雑な組織に有効
機器不要なキットと専用機械用キットの2種類をラインナップ
1日以内でライブラリ作製が完了
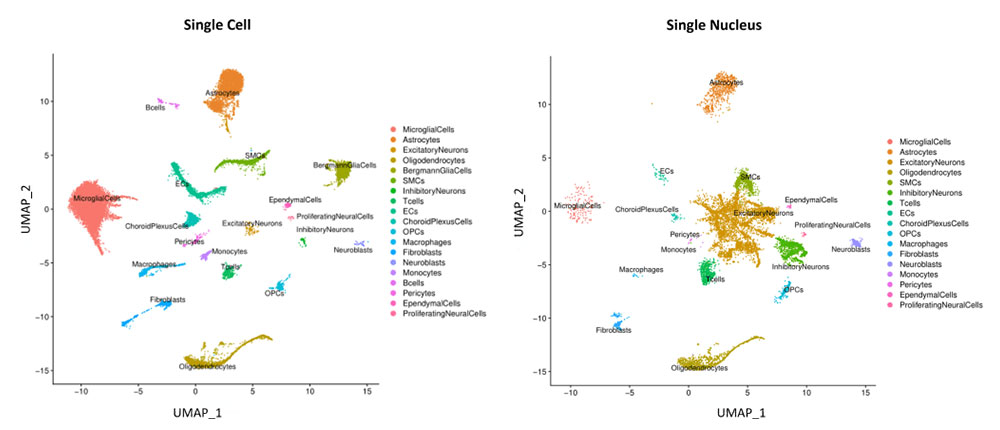
GEXSCOPE Single Cell RNA Library KitとGEXSCOPE Single Nucleus RNA Library Kitを用いたマウス脳組織からのシングルセルシークエンスとシングル核シークエンスの比較。
シングル核データセットでは、神経細胞の集団が顕著に増加していることがわかる。
仕様
| 対応核数 | スタンダードチップ3,000~10,000 HDチップ9,000~30,000 |
|---|---|
| 生成ライブラリ | イルミナ用ライブラリ |
| ワークフロー | 7~8時間 |
GEXSCOPE® Single Cell V(D)J-seqライブラリ調製用マルチオミクスキット
GEXSCOPE® Single Cell V(D)J-seqライブラリ調製用マルチオミクスキットを使用すると、T細胞またはB細胞受容体遺伝子の発現と、シングルセルレベルでの全トランスクリプトーム発現を検出できます。
特長
特許取得済みのSCOPEチップテクノロジーの穏やかで堅牢なマイクロ流体システムを活用して、敏感で希少な細胞タイプでも堅牢な高スループット処理を実現します。
高いRNA捕捉効率により、細胞あたり6k~8KのUMI中央値を取得できます。
多様なBCR/TCRを検出するための強力なV(D)J特異性。
免疫受容体マッピング率の最大化
遺伝子/細胞数の中央値の改善
ビーズ上のカスタマイズされたプローブは、すべてのT細胞/B細胞受容体定常領域を捕捉し、V(D)Jライブラリを構築します。
同時に、同じビーズ上のポリTテールは同じ細胞からのすべてのmRNAを捕捉し、トランスクリプトームライブラリ全体を構築します。
データ例
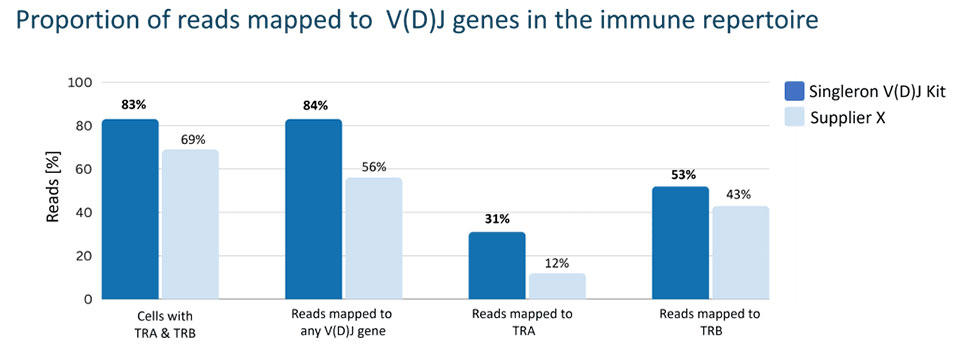
GEXSCOPEシングルセルV(D)Jキットは、転写産物のV(D)J領域を捕捉するように特別に設計されたバーコードビーズにより、他の方法に比べて大きな利点があります。
これにより、V(D)J遺伝子にマッピングされた読み取り数が多くなり、免疫受容体のペアリング率が高くなり、細胞あたりに検出されたV(D)J転写産物固有のUMIの数が多くなり、高い特異性と感度の両方が得られます。
仕様
| 対応細胞数 | 3,000~10,000 |
|---|---|
| 生成ライブラリ | イルミナ用ライブラリ |
| ワークフロー | 14~16時間 |
sCircle® Single Cell全長BCR/TCRライブラリ調製用マルチオミクスキット
免疫プロファイリングのシームレスな精度を実現します。sCircle® Single Cell全長BCR/TCRライブラリ調製用マルチオミクスキットテクノロジーにより、ショートリードシークエンシングでシングルセルレベルのV(D)J領域の全長シークエンシングが可能になります。
特長
cDNA環状化による全長V(D)J配列決定
poly-Aテール捕捉は5’捕捉に勝る
トランスクリプトミクスと全長TCR/BCRプロファイリングを取得します
完全長免疫受容体シークエンシングでは、標準的なシングルセル回収、mRNAキャプチャ、バーコーディングを行い、逆転写の後にcDNAが環状化されます。
cDNAの環状化により、V(D)J領域がシークエンシングプライマーに近づきます。次に、濃縮され断片化されたcDNAを使用して完全長V(D)Jライブラリが構築され、その後、ショートリードシークエンシングを使用して分析されます。
データ例
ハイスループットシングルセルシークエンシングにより、クロノタイプの頻度、BCRまたはTCR転写産物の全長、それらのTRAとTRBまたはBCR IGH/IGLペアリングマップ、およびそれらの可変領域(CDR3)のアミノ酸配列が明らかになります。ここでは、TCRのIGMTV遺伝子とJ遺伝子間の最も頻繁なペアリングが示されています。
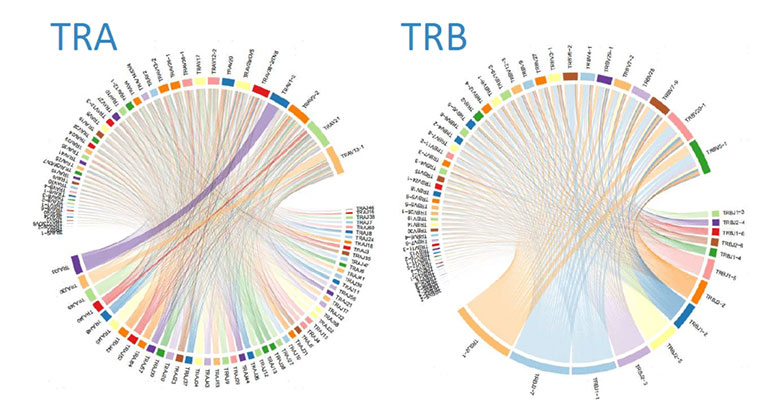 受容体ペアリングマップ
受容体ペアリングマップ
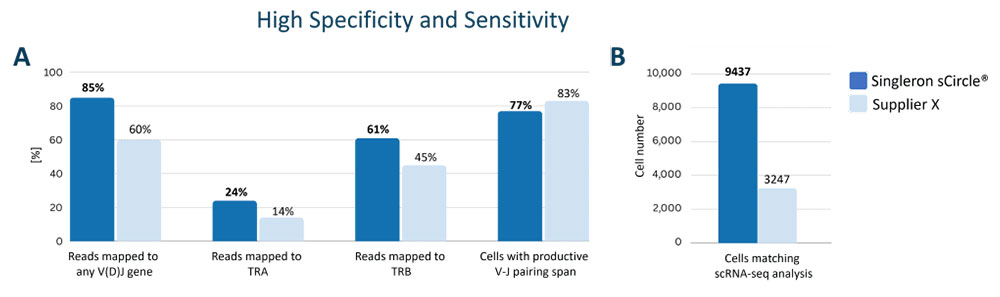
sCircle®シングルセルFull Length免疫受容体ライブラリキットは、転写産物の配列を反転するように特別に設計された免疫受容体cDNAの環状化プロセスにより、他の方法に比べて大きな利点があり、これにより免疫受容体の全長を配列決定できます。
これにより、V(D)J領域と定常領域の一部を含む免疫受容体遺伝子(BCRまたはTCR)の全長配列にマッピングされたリードが得られます。また、免疫受容体のペアリング率が高く、細胞ごとに検出された免疫受容体転写産物固有のUMI数が多いため、特異性と感度の両方が高くなります。
アプリケーション分野
抗体評価
標的治療のための疾患特異的抗体の特定。
自己免疫疾患
免疫システム内の異常なT細胞とB細胞を特定します。
腫瘍微小環境
腫瘍浸潤T細胞およびB細胞を調査します。
個別化治療
免疫療法を開発し、その有効性をテストします。
仕様
| 対応細胞数 | スタンダードチップ3,000~10,000 |
|---|---|
| 生成ライブラリ | イルミナ用ライブラリ |
| ワークフロー | 16~17時間 |
DynaSCOPE® Single Cell動的新生RNA-seqライブラリ調製用マルチオミクスキット (手動用キット)
DynaSCOPE® Single Cell動的新生RNA-seqライブラリ調製用マルチオミクスキットは化学標識を用いて新生mRNAを識別することにより、シングルセル内で新たに転写されたRNAと既存のRNAを区別することができます。
特長
シングルセルレベルでのトランスクリプトーム動態の発見
in vitroおよびin vivoの実験に最適
S4Uラベリング技術に基づくキット
データ例
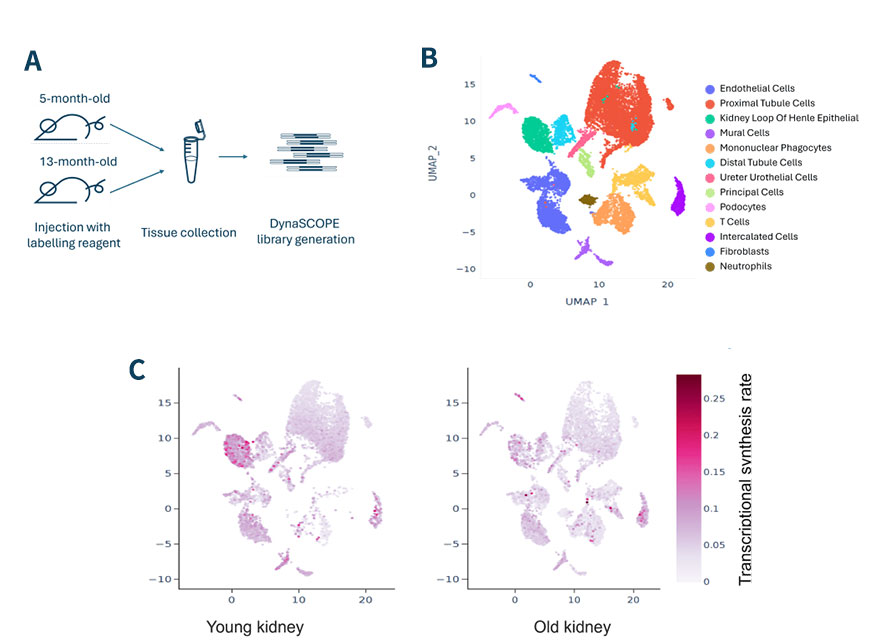 A. 年齢が異なる2匹のマウスに標識試薬を腹腔注射し、DynaSCOPEライブラリー調製を行った
A. 年齢が異なる2匹のマウスに標識試薬を腹腔注射し、DynaSCOPEライブラリー調製を行った
B. 細胞と細胞タイプのアノテーションをUMAPプロットに示す
C. UMAPプロット上に転写合成率を赤色で示す
GEXSCOPEシングルセルV(D)Jキットは、転写産物のV(D)J領域を捕捉するように特別に設計されたバーコードビーズにより、他の方法に比べて大きな利点があります。
これにより、V(D)J遺伝子にマッピングされた読み取り数が多くなり、免疫受容体のペアリング率が高くなり、細胞あたりに検出されたV(D)J転写産物固有のUMIの数が多くなり、高い特異性と感度の両方が得られます。
仕様
| 対応細胞数 | スタンダードチップ3,000~10,000 HDチップ9,000~0,000 |
|---|---|
| 生成ライブラリ | イルミナ用ライブラリ |
| ワークフロー | 9~13時間 |
GEXSCOPE® Microbial Single Cell RNA-seqライブラリ調製用キット (手動用キット)
GEXSCOPE® Microbial Single Cell RNA-seqライブラリ調製用キットは、マイクロウェルベースのSCOPE®テクノロジーを使用して、生物学的酵母サンプルの包括的なシングルセルシークエンス分析を行います。
特長
特許取得済みのSCOPEチップテクノロジーのマイクロ流体システムを活用して、酵母細胞の高スループット処理を実現します。
サンプルあたり最大30,000個の細胞を捕捉
手動または自動処理
単一酵母シークエンシングのための完全なソリューション
データ例
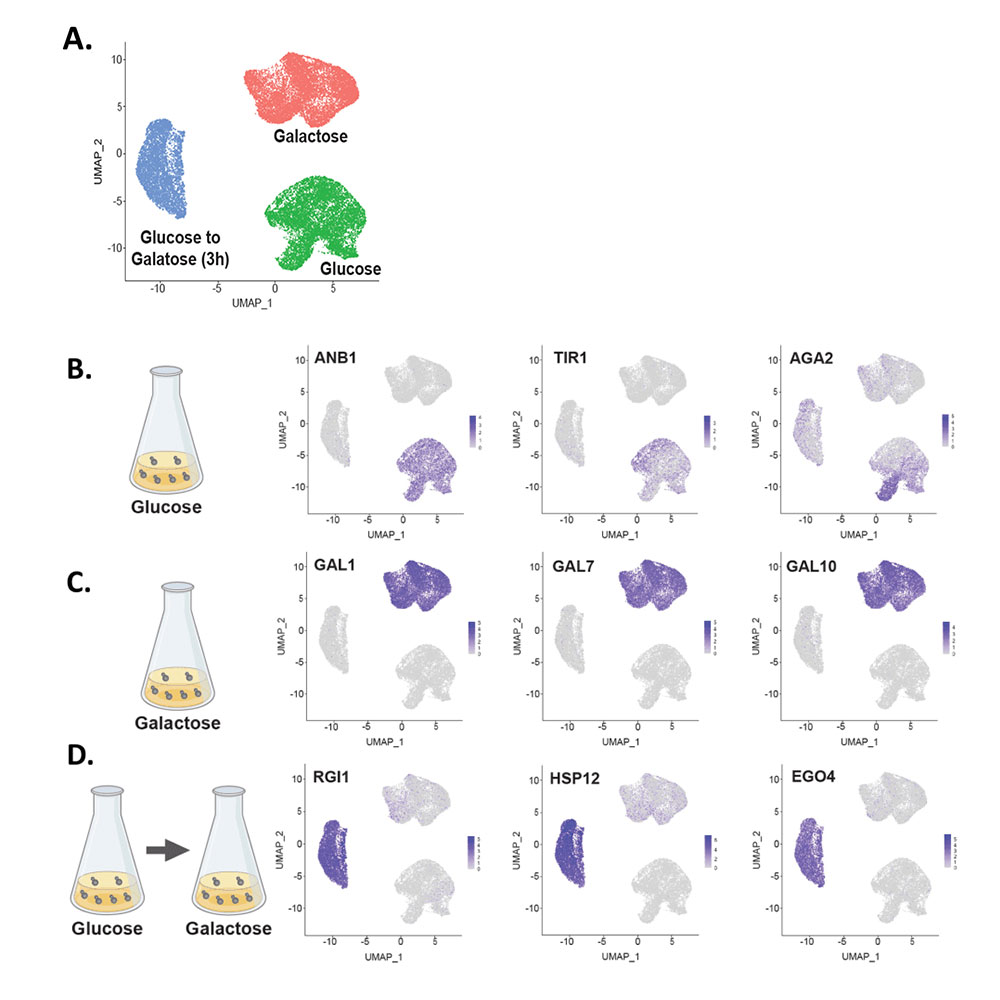
GEXSCOPE® Microbial Single Cell RNA-seqライブラリ調製用キットは、異なる炭素源培地で培養された酵母における異なる遺伝子発現パターンを検出することができます。
(A)グルコースからガラクトースへの炭素源の移行は、酵母において異なる遺伝子発現パターンを誘導します。
(B)グルコースで培養された酵母は、酸素ストレスに関与する遺伝子(ANB1、TIR1、AGA2)の発現を示します。
(C)一方、ガラクトースで培養された酵母は、Leloir経路に関与する構造GAL遺伝子(GAL1、GAL7、GAL1 )を発現します。
(D)グルコースからガラクトースへのシフトは、ストレス応答遺伝子RGI1およびHSP12、ならびにジオーキシックシフトのマーカーEGO4を誘導します。
アプリケーション分野
細胞研究
細胞の状態、細胞周期、生態学的相互作用、集団に関する研究
感染症
ヒト感染症に関与する病原性酵母の研究
細胞工学
細胞のパフォーマンスを最適化するために、発現差のある遺伝子を明らかにする
ストレス反応
ストレスや環境の変化に対する反応を分析する
モデル生物
老化と病気のモデルシステムとしての酵母の研究
仕様
| 対応細胞数 | HDチップ12,000~30,000 |
|---|---|
| 生成ライブラリ | イルミナ用ライブラリ |
| ワークフロー | 7~9時間 |
シングルセルトランスクリプトミクス実験用ツール「Singleron」の
カタログを請求する
無料
お問い合わせ
- お気軽にお問い合わせください。
- TEL:03-6240-0843

